Il DNA di una cellula è organizzato in strutture fisiche chiamate cromosomi. Il cromosoma, o l’insieme di cromosomi, che contiene tutto il DNA posseduto da un organismo viene definito genoma. Nei procarioti, il genoma è generalmente, ma non sempre, un singolo cromosoma circolare. Negli eucarioti è un assetto completo aploide di cromosomi contenuto nel nucleo della cellula (ricordiamo che gli eucarioti contengono anche un genoma mitocondriale e, nelle piante, un genoma dei cloroplasti). E’ importante conoscere l’organizzazione del DNA nei cromosomi per capire il processo mediante il quale l’informazione contenuta in un gene diventa accessibile.
1. Cromosomi virali
I virus sono dei frammenti di acido nucleico circondati da proteine, che infettano tutti i tipi di organismi viventi. A seconda del virus, il materiale genetico può essere DNA a doppio filamento, DNA a singolo filamento, RNA a doppi filamento o RNA a singolo filamento (circolare o lineare). I genomi di alcuni virus sono organizzati in un singolo cromosoma, mentre altri virus hanno un genoma suddiviso in un certo numero di molecole di DNA.

2. Cromosomi dei procarioti
La maggior parte dei procarioti contiene un singolo cromosoma costituito da DNA a doppia elica, circolare. Gli altri procarioti hanno genomi costituiti da uno o più cromosomi circolari o lineari. Generalmente, in questi ultimi vi è un cromosoma principale e uno o più cromosomi più piccoli. Quando un cromosoma piccolo non è indispensabili per la vita della cellula, viene più correttamente definito un plasmidio.

Nei batteri e negli Archei, il cromosoma è organizzato in un blocco denso in una regione della cellula nota come nucleoide. A differenza dei nuclei degli eucarioti, non vi è una membrana tra la regione del nucleoide e il resto della cellula. Se una cellula di E.coli viene sottoposta a lisi blanda, il suo DNA viene rilasciato in uno stato altamente ripiegato. Il DNA a doppi filamento è presente sotto forma di cromosoma singolo e circolare, di 4,6 Mb (mega basi), lungo appossimativamente 1100 μm. La lunghezza del DNA ne cromosoma nel cromosoma di E.coli è approssimativamente 1000 volte la lunghezza della cellula stessa. Quindi, come tutto questo DNA si adatta alla regione del nucleoide della cellula? Il DNA è impacchettato nell’area del nucleoide perchè è superavvolto; in pratica, la doppia elica di DNA è avvolta nello spazio attorno al suo asse.

Per riuscire a capire il superavvolgimento, consideriamo un tratto di DNA lineare di 208 paia di basi nella forma B. In questa molecola vi sono due estremità libere e 20 giri dell’elica, dato che nel DNA nella forma B vi sono 10 paia di basi per giro dell’elica. Se noi semplicemente uniamo le due estremità, produciamo una molecola di DNA circolare. Questa molecola di DNA si definisce rilassata. Alternativamente, se noi dapprima srotoliamo un’estremità della molecola di DNA lineare per due giri e poi uniamo le due estremità, la molecola circolare prodotta avrà 18 giri dell’elica e una piccola regione svolta. Tale struttura non è favorita dal punto di vista energetico e si trasformerà in un’altra con 20 giri dell’elica e 2 giri di superelica (una forma superavvolta di DNA). Il superavvolgimento di per sé produce tensione nella molecola di DNA. Quindi, se viene introdotta una rottura nella molecola di DNA circolare superavvolto, lo svolgimento spontaneo della molecola genera un DNA circolare rilassato. Il superavvolgimento può anche verificarsi in una molecola di DNA lineare. Per una molecola di DNA lineare molto lunga, il superavvolgimento si verifica in regioni localizzate e le estremità si comportano come se fossero fissate.

Esistono due tipi di superavvolgimento, negativo e positivo. Per immaginare il superavvolgimento, si può pensare al DNA a doppia elica come a una scala a chioccola che gira in senso orario. Se svolgiamo la scala per un giro completo, abbiamo lo stesso numero di gradini da salire, ma abbiamo un firo di 360° in meno da fare;, questo è un superavvolgimento negativo. Se invece, avvolgiamo la scala per più di un giro completo, avremo lo stesso numero di gradini da salire, ma un giro di 360° in più da fare; questo è un superavovlgimento positivo. L’entità e il tipo di superavvolgimento del DNA sono determinati dalla topoisomerasi.
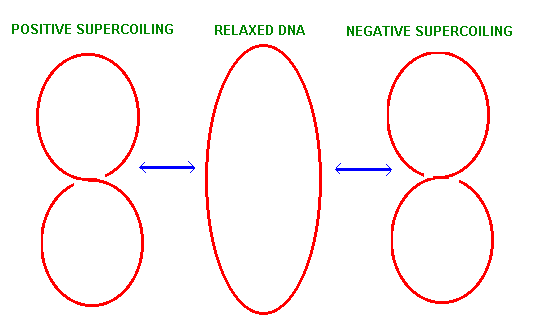
Il compattamento dei cromosomi si verifica anche perchè il DNA è organizzato in domini ad ansa. Nell’E.coli, ogni dominio consiste di un’ansa di circa 40kb di DNA superavvolto, per cui vi sono circa 100 domini per l’intero genoma. Le estremità di ciascun dominio sono fissate, presumibilmente da proteine, in modo tale che la condizione di superavvolgimento del DNA in un dominio non risente degli eventi che influenzano il superavvolgimento del DNA negli altri domini. Il grado di compattamento raggiunto dall’organizzazione in domini ad ansa è di circa 10 volte. Domini ad ansa si trovano in altri procarioti; il numero dei domini è specie-specifico e dipende dalle dimensioni del genoma.
3. Cromosomi eucarioti
Una differenza fondamentale tra procarioti ed eucarioti risiede nel fatto che la maggior parte dei procarioti ha un singolo tipo di cromosoma, mentre la maggior parte degli eucarioti ha un numero diploide di cromosomi in quasi tutte le cellule somatiche (cioè dell’organismo). Il numero di cromosomico umano è 46. Ricordiamo che l’uomo è un organismo diploide (2N), che possiede una serie aploide (N) di cromosomi (23 cromosomi). Ogni cromosoma eucariote è costituito da una molecola di DNA a doppia elica lineare che si estende per tutta la sua lunghezza, complessata con una quantità di peso di proteine circa doppia se confrontata a quella del DNA. Si definisce cromatina il complesso del DNA e delle proteine cromosomiche che costituiscono il cromosoma. La struttura fondamentale della cromatina è essenzialmente identica in tutti gli eucarioti.
Struttura della cromatina
Come abbiamo detto la cromatina risulta costituita da DNA associato a proteine. In linea generale, due tipi di proteine si associano al DNA per formare la cromatina: proteine istoniche e proteine non istoniche. Gli istoni svolgono un ruolo cruciale nell’impacchettamento della cromatina. Una cellula umana, contiene una quantità di DNA 700 volte maggiore di quella del batterio E.coli e senza il compattamento delle 3,4×10^9 paia di basi del DNA nel nucleo diploide, il DNA dei cromosomi di una singola cellula umana sarebbe lungo più di 2,3 metri, immaginando le molecole disposte una in fila all’altra. Diversi gradi di impacchettamento rendono cromosomi, lunghi parecchi millimetri o anche centimetri, capaci di adattarsi entro un nucleo di pochi micron di diametro.
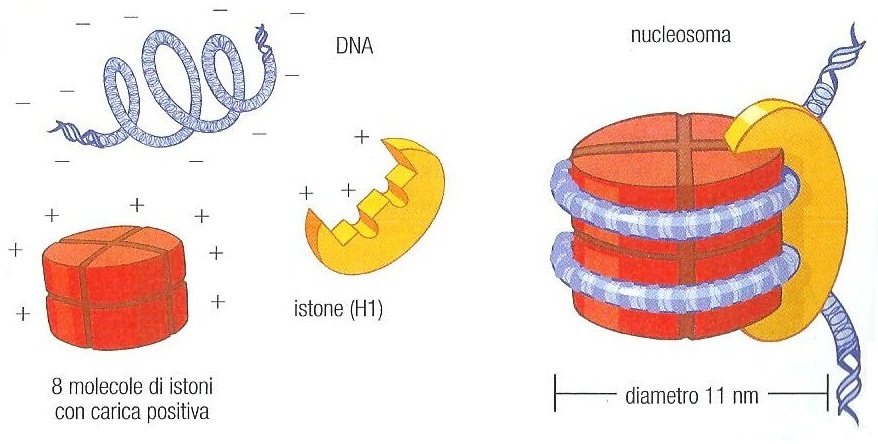
 Il primo livello d’impacchettamento implica l’avvolgimento del DNA attorno a un nucleo di istoni, con formazione di una struttura che viene definita nucleosoma. In un modello del nucleoso, un corto segmento di DNA è avvolto attorno a due molecole per ciascuno dei quattro istoni (H2A,H2B,H3 e H4). Gli istoni del nucleo sono capaci di autoaggregarsi in un ottamero, che è una particella a forma di cilindro appiattito di circa 11nm di diametro e 5,7 nm di spessore. Il DNA si avvolge esternamente attorno al nucleo del nucleosoma compiendo un giro e tre quarti, il che porta a un compattamento del DNA di un fattore di circa sette. I singoli nucleosomi sono connessi da filamenti di DNA linker e da molecole dell’istone H1, noto come istone linker. Le strutture risultanti possono essere osservate al microscopico elettronico come fibre di cromatina di 10 nm (chiamati nucleofilamenti di 10nm) o fibre con un diametro di circa 10nm. Nel suo stato più rilassato, la fibra ccromatinica di un nucleofilamento ha l’aspetto di una “collana di perle”, in cui le perle sono i nucleosomi e il “filo” più sottile che unisce le “perle” e DNA linker nudo.
Il primo livello d’impacchettamento implica l’avvolgimento del DNA attorno a un nucleo di istoni, con formazione di una struttura che viene definita nucleosoma. In un modello del nucleoso, un corto segmento di DNA è avvolto attorno a due molecole per ciascuno dei quattro istoni (H2A,H2B,H3 e H4). Gli istoni del nucleo sono capaci di autoaggregarsi in un ottamero, che è una particella a forma di cilindro appiattito di circa 11nm di diametro e 5,7 nm di spessore. Il DNA si avvolge esternamente attorno al nucleo del nucleosoma compiendo un giro e tre quarti, il che porta a un compattamento del DNA di un fattore di circa sette. I singoli nucleosomi sono connessi da filamenti di DNA linker e da molecole dell’istone H1, noto come istone linker. Le strutture risultanti possono essere osservate al microscopico elettronico come fibre di cromatina di 10 nm (chiamati nucleofilamenti di 10nm) o fibre con un diametro di circa 10nm. Nel suo stato più rilassato, la fibra ccromatinica di un nucleofilamento ha l’aspetto di una “collana di perle”, in cui le perle sono i nucleosomi e il “filo” più sottile che unisce le “perle” e DNA linker nudo.
Nella cellula, la cromatina si trova in uno stato di condensazione maggiore di quella del nucleofilamento a “collana di perle”. Esperimento hanno dimostrato che i nucleosomi possono associarsi fra loro a formare una struttura più compatta, di diametro di 30 nm. Questa struttura più compatta, di diametro di 30nm. Questa struttura è nota come fibra di cromatina di 30nm.
L’avvolgimento del nucleofilamento di 10nm, che produce la fibra di 30nm, condensa il DNA a un sesto della dimensione precedente.
Il livello successivo di avvolgimento implica la formazione di domini di DNA ad ansa, simili a quelli osservati nei cromosomi procarioti superavvolti. La quantità di DNA in ogni ansa va dalle decine alle centinaia di paia di Kilobasi, per cui un cromosoma umano medio ha circa 2000 domini ad ansa. I domini ad ansa si estendono formando un angolo con l’asse principale del cromosoma e sono ancorati ad un’intelaiatura strutturale filamentosa all’interno della membrana nucleare chiamata matrice nucleare.
Le sequenze di DNA associate alle proteine nella matrice sono definite MAR (regioni di attacco alla matrice).
Eucromatina ed eterocromatina
Il grado d’impacchettamento del DNA si modifica durante il ciclo cellulare. Alla metafase della mitosi e della meiosi la cromatina raggiunge il più elevato grado di condensazione. Attraverso la fase G1 dell’interfase, la cromatina gradualmente si rilascia e alla fase S si trova nella condizione di maggior dispersione. In G2 ricomincia la condensazione della cromatina, che arriverà al grado di compattamento elevato della metafase, con il procedere del nuovo ciclo. Visivamente, in un cromosoma metafasico, si osserva che i domini ad ansa si sono ulteriormente avvolti e ripiegati a produrre un cromatidio compatto di 700 nm di diametro. E’ interessante osservare che, se dal cromosoma metafasico vengono rimossi gli istoni, il cromosoma conserva ancora la sua forma generale, con le anse di DNA che sporgono e si estendono da un’impalcatura proteica. I cromosomi interfasici, meno strettamente condensati, hanno a loro volta dei domini ad anse, ma mancano dell’impalcatura proteica presente nei cromosomi metafasici. In base alla colorabilità del cromosoma è stato possibile suddividere la cromatina in eucromatina ed eterocromatina.
- L’eucromatina rappresenta i cromosomi o le regioni cromosomiche che manifestano l’alternanza normale di condensazione e decondensazione durante il ciclo cellulare. La maggior parte del genoma di una cellula attiva è eucromatica. Visivamente, l’eucromatina subisce un cambiamento dell’intensità della colorazione, dal più scuro in metafase al più chiaro nella fase S. L’eucromatina è tipicamente trascritta attivamente, il che significa che i geni in essa contenuti possono essere espressi. Le regioni eucromatiche sono tipicamente prive di sequenze ripetute.
- L’eterocromatina rappresenta i cromosomi o le regioni cromosomiche che generalmente rimangono condensante (con una colorazione più intensa dell’eucromatina) attraverso tutto il ciclo cellulare, anche in interfase. Il DNA dell’eterocromatina spesso replica in ritardo nella fase S rispetto al resto del DNA. I geni all’interno del DNA eterocromatico sono generalmente trascrizionalmente inattivi. Abbiamo due tipi di eterocromatina: la facoltativa e la costitutiva.
L’eterocromatina costitutiva è presente in tutte le cellule in posizione identica su entrambi i cromosomi omologhi di un paio. Questa forma di eterocromatina consiste per la massima parte di DNA ripetuto ed è esemplificata dalle regioni centromeriche.
L’eterocromatina facoltativa, al contrario, varia di condizione nei diversi tipi cellulari, o nei diversi stadi dello sviluppo o a volte da un cromosoma omologo all’altro. Questa forma di eterocromatina rappresenta segmenti di eucromatina condensati e di conseguenza inattivati. Un classico esempio di eterocromatina facoltativa è il corpo di Barr, un cromosoma X inattivato nelle cellule somatiche delle femmine dei mammiferi.
DNA centromerico e telomerico
DNA a sequenze uniche
DNA a sequenza ripetute.
